R-Loop是一种特殊的三链核酸结构,是转录过程中新生的RNA链侵入DNA双链导致形成DNA-RNA杂交双链和一条游离的DNA单链。过去R-Loop被认为是转录过程形成的“副产物”,但是越来越多的证据显示R-Loop不仅广泛存在于原核和真核生物中,也参与了转录调控,基因组稳定性等生物功能,并与多种人类疾病相关。
目前研究R-Loop形成和功能主要是通过特异性识别DNA-RNA杂交体的S9.6抗体或RNase H1,并基于此开发出的多种组学技术,例如DRIP,R-CHIP,Map-R等。有趣的是这些方法在R-Loop的定位和形成上产生了巨大的差异,例如使用催化活性失活RNase H1的R-ChIP和MapR显示出R-Loop主要形成在基因的启动子附近而很少出现在基因的末端,而依赖于S9.6抗体的DRIP等方法则表明R-Loop更多富集在启动子的下游区域以及基因内。这些R-Loop基因组定位方法采用不同的识别因子或捕获策略,目前尚无系统的研究能解决这些R-Loop基因组定位上的差异。
2021年2月17日,武汉大学梁凯威/房萍萍课题组在Science Advances发表题为Genomic profiling of native R loops with a DNA-RNA hybrid recognition sensor 的研究论文,该研究构建了一个新的DNA-RNA杂交体识别因子GST-His6-2xHBD,并结合CUT&Tag技术开发了一种新的R-Loop定位新方法:R-Loop CUT&Tag。基于此技术进一步系统比较了GST-His6-2xHBD和R-Loop识别抗体S9.6,在体外富集和原位捕获定位策略下,获得了R-Loop在基因组上的分布,并且发现不同R-Loop定位方法产生的R-Loop图谱的差异主要由不同的R-Loop捕获策略所导致,而非识别因子本身。

该研究中作者通过串联RNase H1中的DNA-RNA杂交体结合结构域,构建了DNA-RNA杂交体和R-Loop的识别因子GST-His6-2xHBD,进一步通过生化实验证实了GST-His6-2xHBD对DNA-RNA杂交结构的特异性识别能力。随后,作者基于DRIPc-seq的免疫亲和沉淀原理,使用GST-His6-2xHBD替代S9.6抗体进行R-Loop的捕获,结果得到了与S9.6抗体一致的全基因组R-Loop定位特征。为了进一步获取体内真实的R-Loop信号,避免严苛的基因组提取和限制酶消化过程造成可能的R-Loop信号损失,该研究结合GST-His6-2xHBD/S9.6与CUT&Tag技术,通过实验条件的优化和文库构建方法的改进,建立了捕获基因组原位R-Loop的R-Loop CUT&Tag方法。
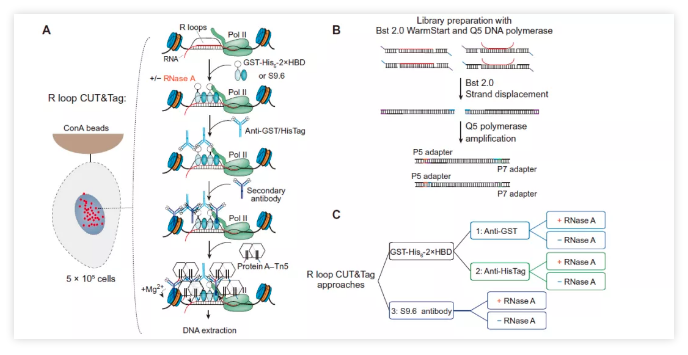
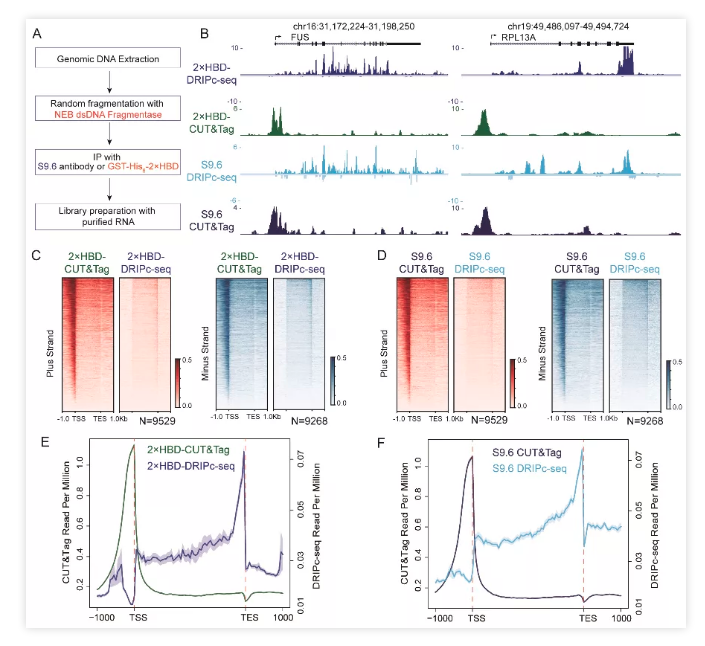
Loop CUT&Tag分析发现GST-His6-2xHBD与S9.6抗体在原位捕获的R-Loop信号非常相似。通过与其他R-Loop定位方法比较发现,R-Loop CUT&Tag不仅能捕获到更加强烈R-Loop信号,而且能捕获到基因间区域和增强子上的瞬时R-Loop信号。该研究进一步引入了随机片段化基因组技术,改良了之前DRIPc-seq方法在片段化过程中的偏好性问题,并且系统地比较了GST-His6-2xHBD和S9.6在DRIPc-seq与R-Loop CUT&Tag定位方法上的差异,发现R-Loop定位上的差异主要来源于R-Loop捕获策略上的差异,而非识别因子本身。
论文第一作者为武汉大学基础医学院硕士研究生王康,博士研究生王红红和李聪慧为共同第一作者。武汉大学基础医学院梁凯威教授和房萍萍副研究员为共同通讯作者,武汉大学生命科学学院陈亮研究员,黄健教授等对该工作有重要贡献。
原文链接:
https://advances.sciencemag.org/lookup/doi/10.1126/sciadv.eabe3516













